Projet METHASEQ : Comprendre le devenir des pathogènes au cours de la méthanisation par mesures génomiques de pointe
Cet article de vulgarisation a été rédigé par Farah Doumit (Club Biogaz , CTBM). Il est basé sur l’étude scientifique menée par :
-
Bonnafous Anaïs, Alvarez-Fraga Laura, Godon Jean-Jacques, Wéry Nathalie (INRAE, Université de Montpellier, LBE, 102 Avenue des Étangs, Narbonne)
-
Moletta-Denat Marina, Azam Olivier, Zennaro Bastien (INRAE Transfert – Metys – LBE – Service Génomique, Narbonne)
-
Laetitia Aubeut-Chojnacki (GRDF, Direction Biométhane, Saint-Denis)
-
Manno Maxime, Bonin Émilie (INRAE Transfert – Metys – Service Génomique, Plateforme GeT-PlaGe, Castanet-Tolosan)
-
Leboucher Anne (Ministère de l’Agriculture, Paris, France)
-
Pourcher Anne-Marie (INRAE, OPAALE, CS 64427, Rennes, France)
-
Adeline Haumont (Association AILE)
-
Chenon Pascale (VoxGaia, Riedisheim, France)
En résumé
- Le potentiel des outils d’analyse des génomes microbiens utilisés en recherche est peu exploité pour la surveillance des méthaniseurs. Le projet METHASEQ (INRAE-GRDF) démontre la faisabilité technique et les avantages de ces nouveaux outils.
- Le devenir de certaines bactéries pathogènes ou indicatrices a été étudié dans cinq unités de méthanisation grâce à trois méthodes complémentaires : culture, dPCR et métagénomique.
- Les résultats montrent que les méthaniseurs agricoles ont une efficacité limitée pour réduire certains indicateurs microbiologiques.
- Un site industriel équipé d’une étape d’hydrolyse acide a, en revanche, montré un fort pouvoir hygiénisant, avec absence de Salmonella, Listeria monocytogenes et Clostridium perfringens dans le digestat final.
- La métagénomique a révélé la présence des pathogènes non détectés par culture ou PCR (ex. Staphylococcus aureus, Listeria monocytogenes), démontrant la nécessité de croiser les approches.
- Ce projet met en lumière la valeur d’une combinaison de méthodes analytiques, offrant une vision plus complète du devenir des pathogènes à l’issue d’une étape de méthanisation.
L’impact sanitaire de la méthanisation, un axe de réflexion légitime
La méthanisation connaît un essor considérable en France. Pour être pleinement acceptée, comme toute unité industrielle, elle doit garantir une bonne maîtrise de ses impacts pour l’environnement et les populations. L’impact sanitaire lors du traitement et de la valorisation de déchets agricoles comme les lisiers ou fumiers ou encore les biodéchets triés à la source, constitue un des enjeux prioritaires de la filière méthanisation dans son exploitation ainsi que dans le devenir du digestat valorisable en fertilisant naturel par application au sol. À ce titre, une question bien légitime se pose : qu’advient-il des bactéries pathogènes présentes dans les intrants au cours des différentes étapes du process de méthanisation ?
Le digestat, sous-produit « matière » de la méthanisation, est épandu sur les sols agricoles dans ses fonctions d’amendement et de fertilisant naturel. Garantir sa qualité sanitaire est donc essentiel, tant pour la santé humaine et animale que pour la protection de l’environnement. Vis-à-vis de la maîtrise du risque microbiologique, les méthodes classiques de suivi reposent encore largement sur la culture microbienne sur milieux sélectifs, mais celles-ci présentent certaines limites pour les matrices environnementales complexes que sont les digestats (flore interférente, sensibilité limitée pour le suivi direct de certains pathogènes). Elles ne permettent pas d’étudier un grand nombre d’espèces microbiennes en une seule analyse, contrairement aux techniques génomiques de dernière génération.
C’est dans ce contexte qu’a été lancé le projet METHASEQ, mené au sein de l’accord-cadre INRAE – GRDF, avec pour partenaires l’Association AILE (Association d’Initiatives Locales pour l’Energie et l’Environnement), VoxGaia, et la Direction Générale de l’Alimentation (DGAL). Son objectif : comparer différentes méthodes d’analyse — culture, digital PCR (dPCR) et métagénomique — afin d’évaluer le devenir des bactéries pathogènes dans les digesteurs.
Pourquoi s’intéresser aux pathogènes dans la méthanisation ?
La méthanisation s’inscrit dans une logique de bouclage des cycles et de valorisation des matières organiques. Pourtant, ces flux contiennent parfois des micro-organismes pathogènes, issus par exemple d’effluents d’élevage ou de biodéchets alimentaires.
Les principaux risques identifiés concernent Salmonella enterica, Listeria monocytogenes, Staphylococcus aureus ou Clostridium botulinum. Cependant, d’autres bactéries pathogènes sont susceptibles d’être présentes, ainsi que des virus ou des parasites. Au vu du grand nombre d’espèces microbiennes qui seraient à rechercher, on utilise des indicateurs comme les bactéries indicatrices Escherichia coli ou Enterococcus spp. On considère que leur mesure (facilitée car leurs concentrations sont plus élevées que celles des espèces pathogènes) permet d’estimer le devenir d’espèces bactériennes proches génétiquement ou dont les conditions de survie dans l’environnement sont similaires.
Les connaissances acquises sur les risques sanitaires en méthanisation concernent principalement les indicateurs et quelques espèces pathogènes. Les méthodes de détection classiquement utilisées reposent sur la culture en milieu spécifique. Ces approches sont reconnues réglementairement, mais elles présentent deux limites :
- Elles se concentrent sur un nombre restreint d’espèces ciblées,
- Elles peuvent sous-estimer les concentrations, certaines bactéries perdant leur capacité à se multiplier dans des milieux de culture dans certaines matrices environnementales.
Dans une logique "Une seule santé", qui relie santé humaine, santé animale et santé des écosystèmes, il est donc crucial de développer une vision plus complète et robuste de la dynamique des espèces pathogènes au cours du processus de méthanisation.
METHASEQ : une enquête scientifique au cœur des digesteurs
Le projet METHASEQ visait à tester de nouvelles méthodes de détection plus sensibles et précises pour mieux comprendre le devenir des bactéries pathogènes.
8 cibles incluant les paramètres réglementaires ont été étudiées
Cette étude porte à la fois sur les indicateurs microbiens réglementaires au titre du respect du règlement sanitaire européen sur les sous-produits animaux (E. coli, Enterococcus spp., Salmonella spp.) et des pathogènes connus pour être présents dans certains intrants (C. perfringens, C. botulinum, Listeria monocytogenes, Staphylococcus aureus et Mycobacterium avium subsp. paratuberculosis).
Sur des sites de méthanisation variés
Les échantillonnages ont été réalisés sur cinq méthaniseurs :
- Quatre unités agricoles, de type mésophile ou thermophile, traitant principalement des effluents d’élevage et des coproduits dont des biodéchets,
- Une unité industrielle intégrant une étape préalable de pasteurisation (70°C pendant 1h) et d’hydrolyse acide (pH<5) en amont du digesteur et ne traitant que des biodéchets.
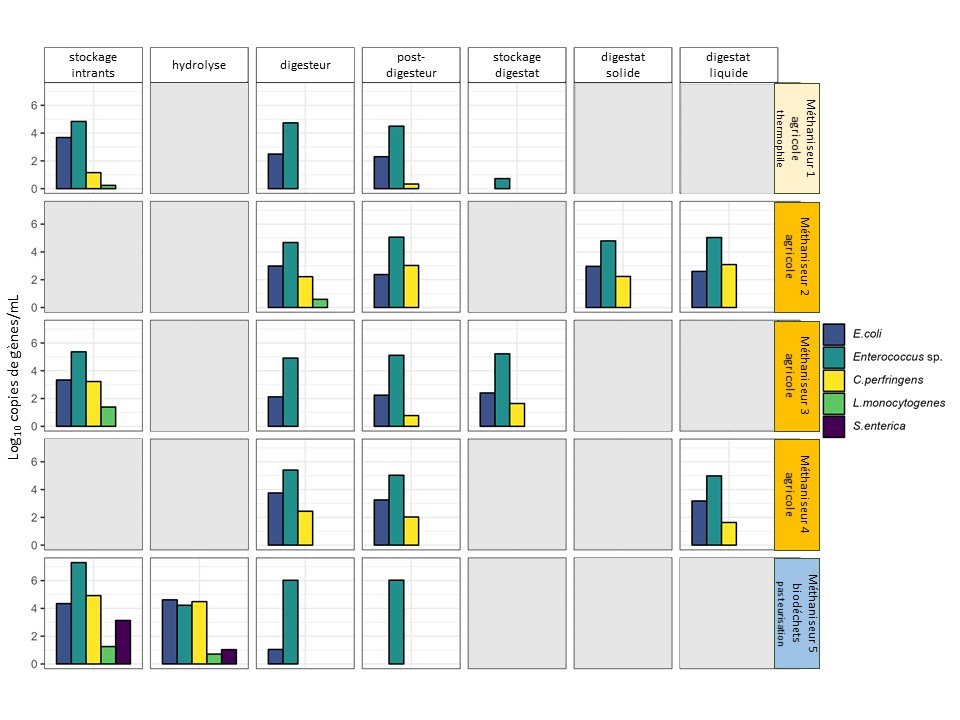
Des prélèvements ont été réalisés à différents stades du processus (cuve tampon, cuve d’hydrolyse, digesteurs, post-digesteurs, stockage des digestats et digestats liquide et solide obtenus après séparation de phase) afin de suivre la dynamique des espèces pathogènes.
Trois approches complémentaires
- Méthodes culturales : utilisées comme référence réglementaire pour dénombrer les bactéries indicatrices et les bactéries pathogènes sur milieu spécifique,
- dPCR (digital PCR) : technique moléculaire permettant de quantifier des gènes spécifiques de bactéries indicatrices ou pathogènes,
- Métagénomique shotgun : séquençage massif de l’ADN présent dans les échantillons, offrant une vision globale de la diversité microbienne.
Cette approche croisée devait permettre d’initier l’évaluation des performances d’assainissement des différentes étapes du process de méthanisation et de comparer la sensibilité des méthodes notamment vis-à-vis des outils moléculaires de dernière génération Le projet a également permis de valider un nouveau protocole d’extraction d’ADN génomique sur des échantillons de la filière méthanisation permettant d’abaisser les limites de sensibilité (1 copie de gène/g), prérequis à l’utilisation de ces outils.
Ce que deviennent vraiment les bactéries dans les digesteurs
L’insuffisance de l’échantillonnage ne permet pas de tirer des conclusions généralisables sur l’inactivation des pathogènes lors des différentes étapes de la méthanisation. Les premières interprétations des résultats obtenus ne sont que les prémices d’une meilleure compréhension de la dynamique des espèces bactériennes où la combinaison de la dPCR et de la métagénomique shotgun constitue une avancée majeure à l’atteinte de cet objectif. Il est important de rappeler également que la présence de pathogène ne signifie pas nécessairement un risque sanitaire et que les concentrations et les voies d’exposition doivent être prises en compte. Parallèlement, l'absence dans un échantillon ne signifie pas absence dans la matière prélevée.
Un effet contrasté entre les sites agricoles et le site industriel
Les premiers résultats montrent une différence nette entre les méthaniseurs agricoles et l’unité industrielle ne traitant que des biodéchets et équipée d’une pasteurisation et d’une hydrolyse acide.
- Dans les méthaniseurs agricoles, les abattements des bactéries indicatrices (E. coli, Enterococcus spp.) restent limités, même dans les digesteurs thermophiles.
- Dans l’unité industrielle, l’hydrolyse acide en amont a eu un effet hygiénisant marqué. Les analyses ont révélé :
- Une réduction significative d’E. coli,
- L’absence de détection de Listeria monocytogenes, Salmonella enterica et Clostridium perfringens dans le digestat final (<1,1 copies de gène /g par dPCR).
Ces premiers résultats confirment que certaines conditions de procédé, en particulier les étapes de prétraitement, peuvent renforcer la maîtrise du risque sanitaire.
Apports de la métagénomique
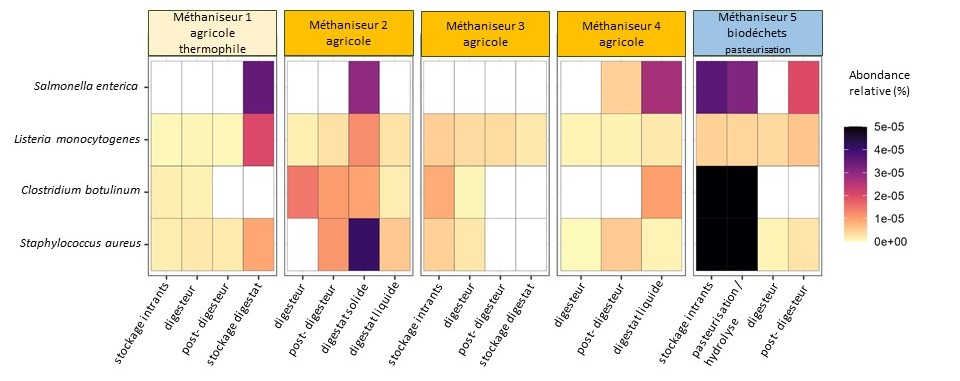
La métagénomique shotgun a mis en évidence une plus grande diversité d’espèces pathogènes dans les biodéchets comparativement aux intrants des méthaniseurs agricoles, notamment Staphylococcus aureus et Clostridium botulinum. En identifiant l’ensemble des entérocoques présents, la prévalence d’Enterococcus cecorum, peu répertorié jusqu’à présent dans les digestats, a pu être mise en évidence.
Cette technique a permis d’élargir le nombre de cibles en une même analyse, ce qui reste difficile par culture ou dPCR. Sa haute spécificité est due au fait que l'ensemble du génome de la bactérie est pris en compte, alors que seul un petit fragment est amplifié par PCR. Cependant, cette technique fournit des abondances relatives au sein de l’écosystème microbien, et non une concentration. Elle est donc complémentaire aux autres méthodes qui sont quantitatives.
Ce constat souligne la nécessité de croiser les approches pour obtenir une image complète.
À plusieurs égards, les informations fournies par les outils moléculaires concordent avec certains faits bien établis qui ont été acquis à l'aide de techniques de culture. Certaines divergences ont été révélées néanmoins (réduction en digesteurs thermophiles), qui devront être explorées.
La combinaison des méthodes, vraie valeur ajoutée scientifique du projet
En comparant les résultats, METHASEQ montre que :
- La culture reste indispensable actuellement pour les analyses normalisées des digestats, même si des normes pour le suivi par PCR quantitative de pathogènes existent dans d’autres secteurs (quantification des légionnelles dans l’eau par exemple),
- La dPCR offre une quantification reproductible et sensible (1 copie de gène/g),
- La métagénomique apporte une vision élargie et permet de détecter des espèces inattendues (ici Enterococcus cecorum). Elle permet également d’obtenir des informations sur le devenir d’espèces en très faible concentration, non quantifiable par dPCR ou par culture (ici Listeria monocytogenes et Staphylococcus aureus).
|
|
Culture |
dPCR |
metagenomique shotgun |
|
Escherichia coli |
4/19 |
17/19 |
15/19 |
|
Enterococcus spp. |
10/19 |
19/19 |
19/19 |
|
Clostridium perfringens |
16/19 |
14/19 |
14/19 |
|
Listeria monocytogenes |
0/19 |
5/19 |
19/19 |
|
Salmonella enterica |
0/19 |
2/19 |
7/19 |
|
Staphylococcus aureus |
0/19 |
0/19 |
16/19 |
Tableau 1 Prévalence des espèces ciblées dans les 19 échantillons analysés par chaque technique
Que retenir de cette étude pour la filière méthanisation ?
Le projet METHASEQ apporte plusieurs enseignements clés pour la filière méthanisation :
- La méthanisation n’élimine pas uniformément toutes les bactéries étudiées. Son efficacité dépend fortement du type d’intrants, du mode opératoire et des conditions de digestion.
- Le procédé industriel intégrant une étape d’hydrolyse ou de traitement acide a montré un meilleur pouvoir hygiénisant. Ces résultats pourraient nourrir les réflexions réglementaires sur les exigences en matière de sécurité sanitaire des digestats.
- La PCR quantitative et la métagénomique doivent être intégrées aux suivis. À mesure que les techniques moléculaires se généralisent dans divers secteurs, tels que la surveillance de la qualité de l'eau et la détection de la contamination microbienne dans l'industrie alimentaire, il est aujourd’hui possible de réévaluer et d'affiner les processus d'inactivation des pathogènes en méthanisation à l'aide de ces mesures génomiques. Une communication scientifique pour plus de confiance existe vis-à-vis des acteurs de terrain. Le projet METHASEQ a démontré la valeur d’une approche intégrée, combinant méthodes culturales, dPCR et métagénomique, pour suivre le devenir des bactéries pathogènes. Plus largement, cette démarche illustre comment la recherche scientifique peut contribuer à objectiver les débats autour de la méthanisation, en apportant des données rigoureuses et transparentes. Elle rappelle donc que la confiance dans la filière repose sur une vigilance continue, au service de la santé publique, de l’environnement et des acteurs agricoles.